Sommaire de la page
Thématique de recherche

Pr Vladimir Torbeev
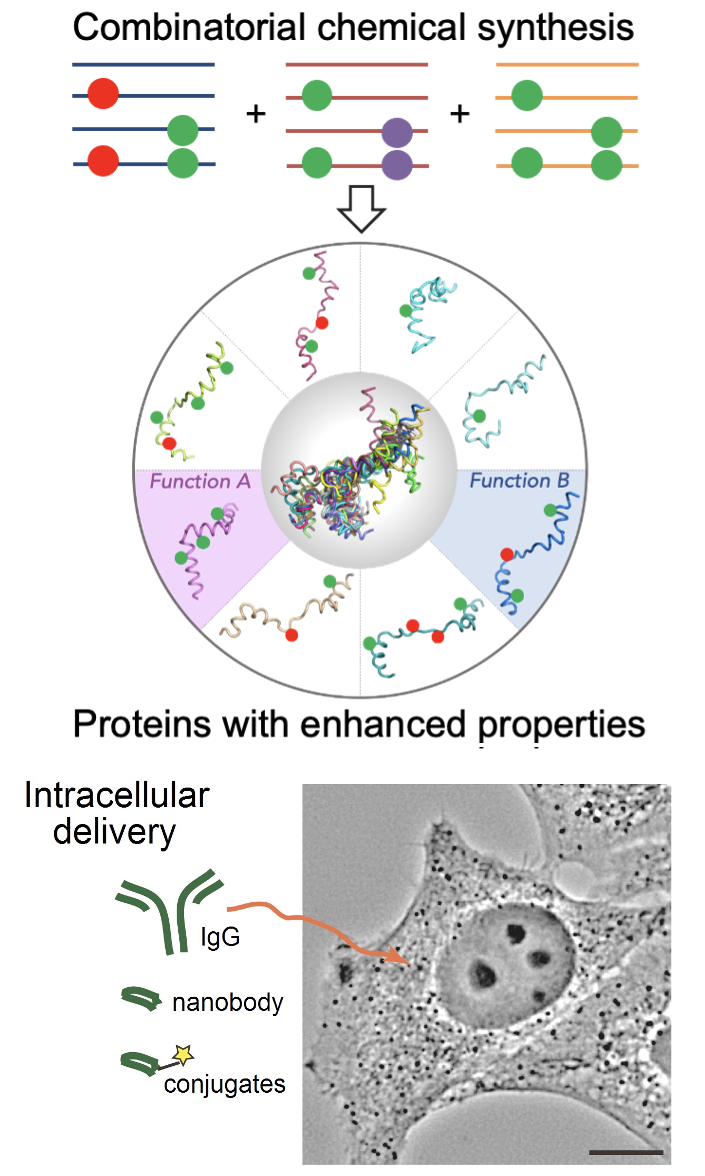
L'équipe de recherche « Chimie des biosystèmes » dirigée par le professeur Vladimir Torbeev est spécialisée dans la synthèse de protéines pour les études biophysiques et biologiques. En combinant la synthèse de peptides en phase solide, l'expression de protéines recombinantes et les chimies de ligature chimiosélectives (par exemple, la ligation chimique native), des protéines uniques peuvent être produites possédant des combinaisons de modifications post-traductionnelles, des marqueurs (isotopiques, fluorescents et autres) ou divers acides aminés non canoniques. Les modifications précisément positionnées facilitent les études structure-fonction, biophysiques et biologiques. Au cours des dernières années, nous avons synthétisé et étudié des oligomères amyloïdes solubles, des variants modifiés des domaines d'activation des corégulateurs transcriptionnels p160 et CBP/p300 ainsi que le domaine de transactivation du suppresseur de tumeur p53. Pour caractériser la fonction des protéines dans les cellules, notre laboratoire développe des approches pour l'administration intracellulaire de biomolécules (supervisé par le Dr Guy Zuber). Les études fonctionnelles et l’activité biologique des protéines synthétiques sont supervisées par le Dr Edwige Voisset.
Protéines intrinsèquement désordonnées. Près d’un tiers du protéome eucaryote est composé de protéines intrinsèquement désordonnées ou de protéines contenant des domaines intrinsèquement désordonnés. Ces protéines jouent des rôles fonctionnels très importants dans la signalisation, la régulation et la reconnaissance cellulaires. De plus, de nombreuses protéines liées à des maladies sont intrinsèquement désordonnées. Nous aimerions mieux comprendre les mécanismes de reconnaissance moléculaire et de formation de complexes impliquant ces protéines, leur rôle dans les réseaux d’interactions protéiques et élaborer des approches pour moduler leurs fonctions à l’intérieur des cellules.
Agrégation des protéines. Les maladies dues au mauvais repliement des protéines (maladies d’Alzheimer, de Parkinson et autres) affectent des millions de personnes dans le monde, cependant, la compréhension actuelle de ces maladies est incomplète pour développer des traitements efficaces. Dans ces maladies, les protéines pliées ou non structurées subissent une isomérisation conformationnelle (mauvais repliement) et un auto-assemblage en oligomères toxiques et en fibrilles amyloïdes. Notre objectif est de décortiquer les détails moléculaires de ces interconversions moléculaires et d'identifier des cibles moléculaires pour la conception de nouveaux médicaments et diagnostics.
Conception de protéines. La synthèse chimique des protéines est très utile pour la conception de protéines avec des structures et des fonctions uniques. Les blocs de construction non canoniques (acides aminés D, résidus fluorés, thiotyrosine) permettent de moduler la conformation, la structure ou la réactivité chimique des protéines. Pour permettre une synthèse hautement efficace de nouvelles protéines, nous concevons des catalyseurs protéiques pour accélérer la ligation de segments protéiques synthétiques et recombinants. De telles ligases artificielles seront particulièrement utiles pour la synthèse de bibliothèques de protéines.
Sondes chémobiologiques intracellulaires. L'administration intracellulaire d'acides nucléiques ou de protéines capables d'interférer de manière ciblée avec la signalisation cellulaire est une clé pour pouvoir moduler les processus biologiques. Les méthodes de vectorisation d'acides nucléiques existantes se sont avérées suffisamment efficaces pour la vaccination ou le traitement de certaines maladies rares. Notre objectif est de contribuer au développement de nouvelles méthodes de transfert de biomolécules à l'intérieur des cellules. Nous travaillons notamment sur le transfert direct de protéines et de sondes chémobiologiques pour suivre et localiser des protéines cibles dans les cellules vivantes en temps réel et à haute résolution.
Contact
Pr Vladimir Torbeev
Soutiens financiers

Membres de l'équipe
- Bureau : D321
- mohamed.banni[at]unistra.fr
- Bureau : D439
- chiper[at]unistra.fr
Hanss Victor
Jouin Alexis
Négroni Luc
- Bureau : D321
- lnegroni[at]unistra.fr
Ofner Shalene
Torbeev Vladimir
- Bureau : D322-3
- torbeev[at]unistra.fr
Trostanetskaia Anastasiia
Voisset Edwige
- Bureau : D322-1
- voisset[at]unistra.fr
Yousef Mo’ath
- Bureau : D321
- Bureau : D306
- zuber[at]unistra.fr
Publications
Iesu L, Sai M, Torbeev V, Kieffer B, Pelta J, Cressiot B (2025). Single-molecule nanopore sensing of proline cis/trans amide isomers. Chem Sci 16(22):9730-9738. doi: 10.1039/d5sc01156f.
Bachelart T, Kumar S, Jouin A, Yousef M, Kieffer B, Torbeev V (2024). Design, Synthesis and Catalytic Activity of Protein Containing Thiotyrosine as an Active Site Residue. Chembiochem 17:e202400148. doi: 10.1002/cbic.202400148.
Grelich-Mucha M, Bachelart T, Torbeev V, Ożga K, Berlicki Ł, Olesiak-Bańska J (2024). Amyloid engineering - how terminal capping modifies morphology and secondary structure of supramolecular peptide aggregates. Biomater Sci 12(6):1590-1602. doi: 10.1039/d3bm01641b.
Aucagne V, Burlina F, Melnyk O, Torbeev V (2021). Des protéines de synthèse taillées sur mesure pour investiguer le vivant. L’actualité chimique 468:26-28.
Sinnaeve D, Ben Bouzayene A, Ottoy E, Hofman GJ, Erdmann E, Linclau B, Kuprov I, Martins JC, Torbeev V, Kieffer B (2021). Fluorine NMR study of proline-rich sequences using fluoroprolines. Magn Reson (Gott) 2(2):795-813. doi: 10.5194/mr-2-795-2021.
Grelich-Mucha M, Garcia AM, Torbeev V, Ożga K, Berlicki Ł, Olesiak-Bańska J (2021). Autofluorescence of Amyloids Determined by Enantiomeric Composition of Peptides. J Phys Chem B 125(21):5502-5510. doi: 10.1021/acs.jpcb.1c00808.
Naudin EA, McEwen AG, Tan SK, Poussin-Courmontagne P, Schmitt JL, Birck C, DeGrado WF, Torbeev V (2021). Acyl Transfer Catalytic Activity in De Novo Designed Protein with N-Terminus of α-Helix As Oxyanion-Binding Site. J Am Chem Soc 143(9):3330-3339. doi: 10.1021/jacs.0c10053.
Bauer V, Schmidtgall B, Gógl G, Dolenc J, Osz J, Nominé Y, Kostmann C, Cousido-Siah A, Mitschler A, Rochel N, Travé G, Kieffer B, Torbeev V (2020). Conformational editing of intrinsically disordered protein by α-methylation. Chem Sci 12(3):1080-1089. doi: 10.1039/d0sc04482b.
Garcia AM, Giorgiutti C, El Khoury Y, Bauer V, Spiegelhalter C, Leize-Wagner E, Hellwig P, Potier N, Torbeev V (2020). Aggregation and Amyloidogenicity of the Nuclear Coactivator Binding Domain of CREB-Binding Protein. Chemistry 26(44):9889-9899. doi: 10.1002/chem.202001847.
Torbeev V, Kent SBH (2020). Chemical synthesis of an enzyme containing an artificial catalytic apparatus. Australian J Chem 73:321-326
Torbeev V (2020). Illuminating Voltage Sensor Paddling in Different Membrane Milieu. Biophys J 118(4):781-782. doi: 10.1016/j.bpj.2019.09.032.
Vergauwe RMA, Thomas A, Nagarajan K, Shalabney A, George J, Chervy T, Seidel M, Devaux E, Torbeev V*, Ebbesen TW* (2019). Modification of Enzyme Activity by Vibrational Strong Coupling of Water. Angew Chem Int Ed Engl 58(43):15324-15328. doi: 10.1002/anie.201908876.
Baral A, Asokan A, Bauer V, Kieffer B, Torbeev V (2019). Chemical synthesis of transactivation domain (TAD) of tumor suppressor protein p53 by native chemical ligation of three peptide segments. Tetrahedron 75:703-708. doi: 10.1016/j.tet.2018.11.074.
Boehringer R, Kieffer B, Torbeev V (2018). Total chemical synthesis and biophysical properties of a designed soluble 24 kDa amyloid analogue. Chem Sci 9(25):5594-5599. doi: 10.1039/c8sc01790e.
Schmidtgall B, Chaloin O, Bauer V, Sumyk M, Birck C, Torbeev V (2017). Dissecting mechanism of coupled folding and binding of an intrinsically disordered protein by chemical synthesis of conformationally constrained analogues. Chem Commun (Camb) 53(53):7369-7372. doi: 10.1039/c7cc02276j.
Ruiz J, Boehringer R, Grogg M, Raya J, Schirer A, Crucifix C, Hellwig P, Schultz P, Torbeev V (2016). Covalent Tethering and Residues with Bulky Hydrophobic Side Chains Enable Self-Assembly of Distinct Amyloid Structures. Chembiochem 17(23):2274-2285. doi: 10.1002/cbic.201600440.
Vergauwe RMA, George J, Chervy T, Hutchison JA, Shalabney A, Torbeev VY, Ebbesen TW (2016). Quantum Strong Coupling with Protein Vibrational Modes. J Phys Chem Lett 7(20):4159-4164. doi: 10.1021/acs.jpclett.6b01869.
Torbeev V, Grogg M, Ruiz J, Boehringer R, Schirer A, Hellwig P, Jeschke G, Hilvert D (2016). Chiral recognition in amyloid fiber growth. J Pept Sci 22(5):290-304. doi: 10.1002/psc.2861.

